1.南京农业大学作物表型组学交叉研究中心 /中英植物表型组学联合研究中心 /江苏省现代作物生产协同创新中心 /现代作物生产省部共建协同创新中心,江苏南京 210095; 2.南京农业大学工学院 /江苏省现代设施农业技术与装备工程实验室,江苏南京 210095; 3.国家信息农业工程技术中心 /农业农村部农作物系统分析及决策重点实验室 /智慧农业教育部工程研究中心 /江苏省信息农业高技术研究重点实验室,江苏南京 210095; 4.数字科学研发部,英国国立农业植物研究所 /剑桥作物研究中心,剑桥 CB3 0LE,英国
摘要: 植物表型组学研究正逐渐向综合化、规模化、多尺度和高通量的方向快速发展。本文首先介绍了植物表型研究的最新动向。然后针对室内表型监测平台的特点和各类室内表型针对的表型性状进行了系统介绍,包括产量、品质、胁迫抗性(包括干旱、抗冷热、盐胁迫、重金属和病虫害)等。在此基础上,本文还根据通量、传感器集成度和平台大小等把一些国内外流行的室内植物表型平台进行了分类,并介绍了这些室内表型平台在植物研究中的应用情况。同时,本文还介绍了室内表型数据的管理和解析方法。最后,本文着重讨论了室内表型平台的发展方向,并结合中国植物研究的实际情况对表型组学在中国的发展提出了展望,以期为中国植物表型研究提供指导和建议。
关键词: 植物表型组学;室内表型监测;产量性状;品质性状;抗性表型;表型数据管理和解析分类
中图分类号: S-1 文献标志码: A 文章编号: 202003-SA002
引文格式:徐凌翔, 陈佳玮, 丁国辉, 卢伟, 丁艳锋, 朱艳, 周济. 室内植物表型平台及性状鉴定研究进展和展望[J]. 智慧农业(中英文), 2020, 2(1): 23-42.
Citation:Xu Lingxiang, Chen Jiawei, Ding Guohui, Lu Wei, Ding Yanfeng, Zhu Yan, Zhou Ji. Indoor phenotyping platforms and associated trait measurement: Progress and prospects[J]. Smart Agriculture, 2020, 2(1): 23-42.
1 植物表型组研究发展现状
近年来随着跨学科植物研究的深入,植物表型组学的重要性和迫切性已获得了学术界的普遍认可,例如作物育种可以通过高通量表型采集和分析技术推动基因分型和基因组研究[1,2],植物种质资源的构建需要依靠表型组学扩展新群体,快速寻找重要性状和总结作物改良途径[3-5],表型分型技术可以促进基因资源库和遗传多样性的有效管理[1,6,7],大幅提高作物栽培效率,相关理论积累也需要通过对重要农艺性状的自动化提取和快速分析完成[8,9]。此外,田间表型大数据的高速获取和有效管理还有助于农业生产的精准化和可持续发展等[10,11]。因此,植物表型组学不但可以为基础研究服务,还可为作物育种、栽培和农业生产等应用研究提供理论基础和技术手段。植物表型的监测和分析手段也逐渐向综合化、规模化、多尺度和高通量的方向发展[12,13];表型信息的获取从原来的单一时间点、人工观测为主的方式,正朝全生命周期、多尺度和自动化的方向改进[5,14]。
目前,欧美主要发达国家的政府和研究机构都在针对植物表型研究积极布局,对该领域投入了大量的时间和经费来建设可共享的室内外植物表型平台,而且还出台了多项国家级的研究计划,例如由欧盟研究理事会资助的欧洲植物表型联盟(European Plant Phenotyping Network, EPPN 2020;www.plant-phenotyping-network.eu)和旨在共享欧洲各国表型设施的EMPHASIS计划(emphasis.plant-phenotyping.eu),已持续超过10年的澳大利亚植物表型研究平台计划(Australian Plant Phenomics Facility;www.plantphenomics.org. au),英国最新设立的国家植物表型组计划(PhenomUK,www.phenomuk.net),由美国国家科学基金会和能源部共同资助的TERRA-REF平台计划(terraref.org/data/sensing-components),由瓦赫宁根大学和乌得勒支大学共同承建的荷兰植物生态表型研究中心(Netherlands Plant Eco-phenotyping Centre;www.wur.nl/en/product/The Netherlands-Plant-Eco-phenotyping-Centre-NPEC.htm),以及由民间发起、德国尤利希研究所牵头的国际植物表型联盟(International Plant Phenotyping Network,IPPN;www.plant-phenotyping.org)
[16-19]。上述国家级大型科研项目一般以建设多尺度表型数据采集和分析平台为主,通过室内外表型获取技术采集各个层面(如细胞、组织、器官、植株和群体等)的植物表型组数据,再使用各类传统或智能化数据解析方法从海量数据中提取可信度高、高可重复性的重要性状信息[11,13],进而为解决基因挖掘、作物育种栽培以及农业生产等中的实际问题提供大数据支持和方法学支撑[20-23]。
中国植物界对表型组学的研究热情也呈持续上升的趋势。根据不同的应用场景和研究方向,中国植物表型组学研究在植物监测技术[24]、作物育种[25]、种质资源鉴定[26]、农业生产[27]、田间作物表型信息获取和解析[28-31]、作物精确栽培应用[32-34]、平台建设和技术应用[35]、农业大数据管理和挖掘[36]研究领域的进展[37,38]及表型研究现状和展望[22,39]等多个方面取得了显著的进展。
2 室内表型监测平台应用场景和主要特点
表型数据采集和分析手段是植物表型组学研究的核心部分,包括了室内和室外两部分。根据平台和设备的不同,室内外采集技术大致可分为人载(包括手持)、车载、定点监控、大型自动化平台和室外航空机载及卫星成像等;其数据采集和管理具有数据量大、附加信息多(如地理位置、光温水气等环境因素)、获取标准不统一、数据不确定性高、重复性低和时效性高等多个特点[39-41]。如何通过大数据分析对采集的植物表型组数据进行预处理、在整合多源数据和数据挖掘技术的基础上把海量数据转化为可用的性状信息是现在表型组学所面临的最大挑战。由于室内表型监测技术可以对各类环境因素进行精确调控(如在人工气候室对空气温湿度、光照长度和光谱区间、以及相应的水、热、光周期等进行调节),同时还可以严格控制生长箱或温室中的植物的生长条件(如对土壤中水分和养分的监控、种子原生态环境条件的控制等),因此室内表型监测技术能够在复杂实验条件下对作物生长发育进行精确分级模拟和针对性研究[8,18]。基于室内表型平台精确调控、分级模拟和自动化精准采集等特点,室内表型检测有着室外表型技术难以复制的优势,因此其在植物研究中有着重要的意义。本文将着重对室内表型检测平台技术及相应的性状鉴选问题展开讨论,即在不同室内研究条件下,根据不同的植物研究目标,通过室内表型获取和后期数据解析完成对植物关键性状的鉴定。
现代化室内高通量植物表型检测平台一般通过自动化传送设备和集成化传感器(如可见光、近红外、远红外、荧光、多光谱、激光、高光谱等)完成对与植物遗传与变异等密切相关的植物动态生长发育表型数据集的获取。其优势为高分辨率、高可控性和高通量,可为后续的植物表型分析提供高质量的多维图像和实验数据[4,42,43]。无论是地上还是地下部分,室内表型监测在尺度上通常以单株为单位,通过归集单株特征描述群体特征。根据图像传感器精度,所提取的表型数据往往可以精确到组织细胞层面。因此,室内表型性状的尺度划分可上至群体,下至组织细胞。比如,在细胞层面上,如图1(a)所示,通过高速共聚焦显微镜(High-speed Confocal Microscopy)和高通量细胞筛选系统(High-content Screening System)可对大量细胞表型开展植物抗病遗传机制等方面的研究,包括胞吞作用(endocytosis)[44],胼胝质沉积(callose deposition)[45]和细胞结构变化的表型分析[46]。在组织和器官层面,如图1(b),多种自动或半自动的室内成像系统可获取各类锈病、发芽率和花期等图像光谱数据,以此开发计算机图像分析算法提取重要表型特征参数[47-50]。在植株层面,图1(c),自动化传送系统和多角度、多传感器的拍摄技术可对盆栽植株进行图像获取和近实时植株分割[51,52],以此完成对多植株的连续生长性状监测[49,53]。在大型室内环境中还可以对小群体进行表型数据采集和表型分型鉴选(图1(d)),如在温室中使用经济型激光雷达(LiDAR)对小群体进行三维重建,进而分析作物产量性状[54],以及通过集成多光谱和高清成像设备高通量采集室内种植的作物小区的关键农艺性状等[5,8]。值得指出的是,很多自动化室内表型平台和设备的研发皆由商业机构和大型科研机构推动,其中包括德国LemnaTec的高通量室内植物表型筛分系统(如LabScanalyzer和Scanalyzer HTS),捷克PSI的PlantScreen系统,比利时CropDesign的TraitMill系统(已被德国巴斯夫集团收购),华中农业大学和华中科技大学共同开发的水稻表型设施HRPF,以及中国科学院植物研究所和上海泽泉科技股份有限公司共同研发的PhenoWatch系统等。这些系统被广泛应用于室内植物的统一管理和可控条件下的植物形态特征的监测,应用于突变体筛选
[55],抗旱性分析[4],生长表型和产量的相关性分析[8],水稻全基因组关联研究和基因座定位[5]及作物高精度三维重建和表型结构参数的提取[54]等不同研究方向。
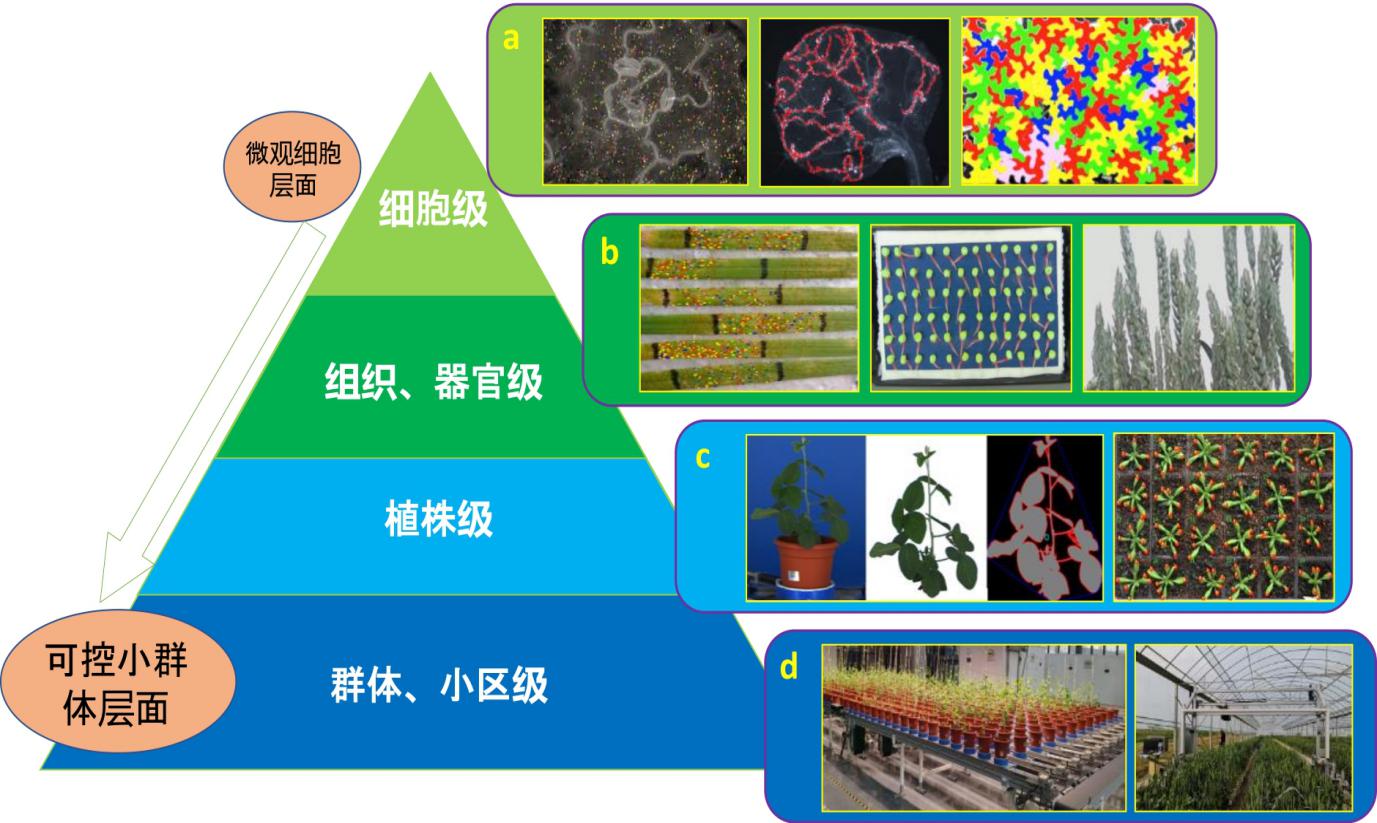
(a)细胞层面对胞吞作用[45],胼胝质沉积[46]和细胞结构变化的表型分析[12]; (b)组织和器官层面对各类锈病、发芽率和花期等性状分析[47-50];(c)单株层面对单株的图像获取、植株分割和生长监测[49,51-53]; (d)小群体和小区层面的表型数据采集和关键农艺性状分型鉴选[5,8,54]。
图1 不同层面的室内植物表型监测及其分析对象
Fig. 1 The scope of multi-level indoor plant phenotyping and associated phenotypic traits
通过结合室内环控设备(如可控温室和人工气候室),室内表型采集技术还可以模拟多种野外生长条件,进而评估表型的可塑性和稳定性,全方位对关键表型性状(如产量、品质和各类抗性指标等)进行鉴定,获得具有统计学意义的研究结论[42,56,57]。由于这类研究很难在室外通过调控田间试验条件完成,因此室内表型监测在数量性状基因座(Quantitative Trait Locus,QTL)分析、全基因组关联分析(Genome-wide Association Study,GWAS)、基因表达鉴定等方面有显著的优势。室内表型监测是最早应用于品系筛选、关联表型和基因组信息及遗传结构解析等研究方向上的植物表型技术[20,58,59]。最近几年,通过综合表型组信息、主要环境因素和遗传基础,室内表型监测还逐渐被应用于建立各类产量、品质和抗性模型,以此预测不同作物与重要环境因素动态互作下的表现[8,13,60]。
3 室内表型监测平台所针对的表型性状
在过去的十年中,植物表型组学已逐渐从新兴研究课题发展成为高速发展的多学科交叉研究领域。表型作为基因型和多种环境间互作的结果(Genotype-environment Interaction,GxE),不仅可以通过细胞、组织、器官和植株的物理结构变化来衡量,还可以通过各类生理生化相关的表型参数来验证[7,61]。国内外各类室内植物表型平台一般基于上述思路进行设计研发。从比利时CropDesign公司的第一套大型植物高通量表型分析平台TraitMill问世至今[62],室内植物表型性状获取技术正向着多样化、自动化和高通量的方向发展。其获取的性状可分为地上(如生物量、株高、穗粒数和结实率等收获指数)和地下(如二维、三维的根系形态性状等)两部分[42]。根据不同的生物学问题,本节将着重介绍室内表型平台所针对的各类表型性状及其相对应的采集参数和生物学问题。
3.1 产量相关表型性状
产量的本质是生物量,作物产量与收获植物器官如禾谷类作物种子的生物量有极显著的相关性[63]。对产量评估一般包含三个重要指标:单位面积穗数,每穗粒数和籽粒的单位重量[64]。由于这些指标很难在田间试验中无损检测,因此与产量相关的表型被大量应用于各类作物产量检测和研究中[65]。在室内条件下可检测的与产量相关的表型性状分为地上和地下两部分:地下部分针对植物根部的根系尺寸(如根深、二维根宽和根长等)和根系的三维形态、结构特征等[66-68];地上部分针对一些重要器官的形态参数(如叶长、叶宽、冠层面积、植株高度和三维植株体积)和一系列重要农艺性状(如分蘖数、叶数及单位面积穗数等)[21,69,70]。最新研究还借助X射线断层扫描分析技术(X-ray Computed Tomographic Analysis)对麦穗和穗粒分别进行三维特征重建,以此完成不同处理条件下穗重、粒重和粒数与粒型和穗型的相关性分析[71]。产量相关性状与植物生长发育密切相关,因此各类生长特性曲线(即生物量和时间的相关性)也被用于产量评估,比如在室内表型研究中会使用生长速率结合生物量参数对作物产量做相关性分析[72],通过多角度摄像和多视图运动恢复结构技术(Structure from Motion with Multi-View Stereo,SfM-MVS)获取盆栽小麦的株高,叶数和分蘖数等特征,最后通过人工校验得出作物早期生物量和叶面积的增长率与最终产量的关系[60]。除了形态结构,产量也可通过与光合作用相关的表型参数来预测[73]。在不同光照强度下,通过表型性状可评价各项光能转化指标,以此在植株层面对生物量的换算进行建模。这一类分析方法还被应用于光能、营养、CO2释放速率、水分利用效率和单株产量的精确检测分析[74]。
收获后的产量表型至关重要,因此如何在收获后提取可靠的产量表型性状也获得了研究者们的重视。比如种子收获后通过phenoSeeder平台的机器臂和吸盘可对单个大麦或油菜等种子进行单个籽粒的三维形态分析和称重
[75]。因为对禾本科籽粒的长宽、数量和重量进行大规模自动化分析可准确反映产量,所以新型的玉米籽粒性状测定系统(Maize Kernel Traits Scorer,MKTS)通过线扫描成像、图像处理和自动控制技术自动测量玉米籽粒的12种性状,为玉米遗传和玉米育种研究提供可靠的产量相关参数[76]。微型计算机断层扫描技术(micro-CT或µCT)近来也被应用于产量分析,在对玉米籽粒之间的差异和玉米丰满度(如籽粒密度,孔洞,孔隙率等表型)进行分类评价后,综合这些参数可反映玉米产品的产量和质量,为鉴定玉米的品质和种质资源服务[77]。
3.2 品质相关的表型性状
室内条件下与作物品质相关的性状一般围绕植物收获器官的形态结构变化和生理生化指标等展开。以禾谷类作物为例,各类生理生化指标一般通过高光谱(如近红外)拍摄的光谱照片结合多光谱分析技术无损检测稻麦或玉米的营养成分如直链淀粉、葡聚糖等碳水化合物[78,79]。收获器官的油脂含量、蛋白质含量和水分等也可得到相对准确的测量[80];相同的方法也被应用于检测植物中的各类微量元素(如铁、锌、硼、磷等),以此评估作物品质[79,81]。此外,结合高光谱反射和荧光成像系统的摄像设备还被用于分析各种化学成分在植物组织中的空间分布情况(包括油脂、蛋白质、水分和碳水化合物等),为作物根据营养成分分级提供量化标准[82]。
值得指出的是,作物品质通常很难仅仅依靠形态结构特征来评价。因此,结合收获器官的各类营养含量和品质相关的形态特征(如收获器官的大小、生长速率和颜色变化等)亦可作为品质分级的依据[79,83]。比如,对各种园艺作物的形状、大小、色泽、种皮厚薄、纤维长度和强度等性状分析可以对产品的综合品质加以定义[84,85]。最后,近年来随着代谢组学的发展,越来越多的小分子化合物也能被检量,通过化合物组成可定位代谢相关的基因组,也为品质检测提供了新的途径[81,86,87]。
3.3 胁迫相关的抗性表型
生物和非生物胁迫(biotic and abiotic stresses)可导致产量和品质的下降,是植物表型研究的重要方向之一[21]。在室内条件下,对各类逆境模拟技术已日趋成熟,可针对不同作物设置不同逆境和胁迫实验,以此研究环境因素对植物生长发育的影响,为抗性鉴定和农业生产提供指导[10,88]。在全球气候变化加剧和人口压力渐增的今天,如何在恶劣天气条件下维持作物产量和品质变得愈发重要。因此高通量、大规模鉴选具有明显抗性特征的作物品种将加速作物遗传选择和选育栽培的研究进程[89,90]。植物抗性可分为避性、御性和耐性三种形式[14,91],根据植物与不同逆境的交互情况,又可分为抗旱、抗涝、抗冷(抗冻)、抗热、抗盐、抗污染、抗病等特性[92-94]。本节简要介绍了室内平台可测的抗旱、抗冷热、抗盐、抗污染(重金属)和抗病虫害等数种抗性特征。
3.3.1 干旱胁迫
中国因干旱造成的粮食损失一度达到每年数百亿元人民币[95,96],利用表型组学技术加速作物抗旱性状的研究有迫切的现实意义。在温室培养下,Chen等[4]使用基于可见光、荧光和近红外等多种光谱技术对18个大麦基因型的400多个性状进行了统计;然后通过多重共线性分析(Multicollinearity Analysis)等方法筛选出六个主成分性状簇,以此分析作物对干旱的敏感程度。前文提到的X射线断层扫描分析技术对麦穗和穗粒的三维分析[71],其实验设计也是为了研究不同干旱胁迫下穗重、粒重和粒数的变化。针对水稻抗旱育种,Guo等[97]基于图像提取了507个水稻品种的51个基于图像的性状(i-traits),并通过这些i-traits监测不同水稻品种的干旱反应、性状的遗传力和变异,进而通过全基因组关联研究确定了470个关联基因座。
基于根发育和根形态的表型组学研究对于研究作物抗旱机理同样作用显著。这类研究一般将根系浸置于琼脂中或半悬空拍摄,因为干旱胁迫能够阻止次生根的发育,可将悬空的根系一侧贴在湿滤纸或琼脂上可形成水势梯度,使次生根的分生组织向有水的一侧发育[98,99]。在根袋中培育幼苗获得幼根的构型也被用于抗旱性的筛选,通过总根长和侧根数的计量可分析抗旱与产量的关系[67]。生长素、脱落酸和细胞分裂素等植物激素对根形态发育起到重要作用,因此有的研究团队对干旱条件下根系中的特定激素也开展了一系列研究[100]。较为昂贵的核磁共振成像(Nuclear Magnetic Resonance Imaging,NMRI )技术也逐渐被用于对植物组织中水分的鉴定,借此完成对抗旱性的精确分级[101,102]。
3.3.2 抗冷或抗热胁迫
用红外线获取水稻冠层温度对高温胁迫的响应,可发现热胁迫下冠层温差大的品种有更好的耐热性(canopy temperature depression),这是因为蒸腾作用有冷却效果
[103],通过这种方式获取的材料可作为潜在的耐热种质资源。此外,通过3D图像可以测得热胁迫下番茄的生物体积(biovolume,一种生物量的度量方式),从而检验和筛选耐热基因型[104]。和其他胁迫类型相比,与冷胁迫相关的表型组学研究主要集中于检测叶绿体荧光变化和光合作用的关系[105]。拟南芥在经过4℃低温驯化(acclimation)后投入到寒冷环境中(10、4、0和-1.5℃),驯化后的材料光合作用峰值提前,荧光量子产额(Quantum Yield of Fluorescence)中表征非光化学淬灭的比重增加,光合作用比重减少[106]。通过荧光图像获取的表型数据也可用于对部分水稻抗寒品种从低温环境回归理想环境后的光合作用进行表型分型[107]。
3.3.3 盐胁迫和重金属胁迫
干旱环境在植物生理上存在渗透压胁迫,这也是盐胁迫所产生的生理效应[108]。由于可耕种土地的日益减少和围海造陆策略的推广,盐碱地和滩涂地的利用使得作物抗盐的重要性与日俱增。生理方面,盐胁迫除了和干旱一样存在渗透压胁迫外,还有离子胁迫,而且后者对叶绿体影响较大。因此,可以用叶绿体荧光光谱特征来区分早期的渗透和离子胁迫,以此研究作物在盐胁迫下的生长发育情况[109]。此外,根据荧光波段由红向绿的规律还可通过谱带制作数码标记来指示离子胁迫水平,标注盐胁迫下的早期生长速率。当然,幼根在盐胁迫下也存在一定的避性,即幼根有向着盐浓度低的方向生长发育的表型特征[110]。
重金属胁迫与盐胁迫机理相似,在琼脂中建立不同重金属离子的浓度梯度可对胚根表型进行分析,胚根会根据浓度产生生长方向改变及增粗的表型特征[111]。重金属等有毒有害物质除了影响作物产量外还会影响作物的卫生品质,即污染物会从根部向上迁移影响到收获器官。当前污染物在作物体内追踪主要依靠X光技术[112],通过X射线荧光光谱仪可探测菜豆种子中的锌含量、桉树根中的铅含量,以此获取污染物原位分布图和含量评估[113]。依靠X射线吸收结构荧光还被用于获取水稻和小麦中砷在各器官中的含量,可判断砷是否在地上部分富集[114]。
3.3.4 病虫害胁迫
作物对病虫害的抗性不同于对非生物胁迫的抗性,存在着寄生或取食等种间关系。因此植物抗病虫害的等级一般以其被寄生或取食的表型来评判,如叶片受病虫害入侵的程度,病斑数量和大小、病斑颜色和病叶比例等[115,116]。病害等级能够通过红外光谱成像来获得叶片病斑面积,通过人工校验可以区分病斑和衰老造成的斑点[117,118]。通过深度学习可以对大量植物疫情照片训练,建立模型后对疫病的种类(如斑病、锈病)和程度进行准确鉴定[119]。热红外感光也有通过温度指示疫情的研究实例,例如花生接种叶斑病后经测算叶片温度较健康叶片高2~3℃[118]。和抗冷、抗热胁迫一样,植物病虫害表型主要是对胁迫的响应,包括功能受损和防御策略:如大麦抗粉病品种Irina的病斑可表征抗病反应[120];在病原体攻击时植物抗病基因产生的病斑数可表征抗病等级[121];害虫的侵入也可以通过连续监测害虫的行为来分析,如蓟马在拟南芥叶片上的停留时间被用于评估植物抗性的依据[122]。
与害虫的取食不同,病原菌更加微观,寄生于胞内或胞间。因此,植物的抗性还可通过更加微观的组织或细胞表型来反映。例如通过显微镜或高通量细胞筛选系统可获得胞间物质传递或胞内细节的荧光图像,再通过图像处理实现细胞尺度的抗病表型分析[123-125]。对病原体鞭毛诱导特异性受体抗病蛋白后产生的荧光拍摄,可以确定这种蛋白跨膜运输的激活机制[125];细胞自噬本身不像受体那样能识别病原体蛋白,其抗病作用可通过分解自身能量贮存物或分解病原体来实现,因此识别自噬结构也是抗性鉴定的重要环节[126];微管蛋白的形成是另一种非特异性的抗逆机制,同样可以依靠图像处理实现细胞表型的定量分析[46];在不依靠荧光的情况下,通过高通量拍摄气孔的闭合程度亦可分析植物抗旱和抗病机制[127]。
4 室内植物表型监测平台分类和数据解析方法
上文所述的各类表型性状需要多种技术手段采集和解析,因此多种室内表型获取平台得以开发。由于室内植物表型平台的分类在国际上没有统一标准,对于平台的划分一般根据通量、价格、传感器集成度、平台规模大小和占地面积等方面综合考量。本文在综合了室内植物表型平台的测量对象和规模后,将室内平台分为大中小3类进行介绍(图2)。
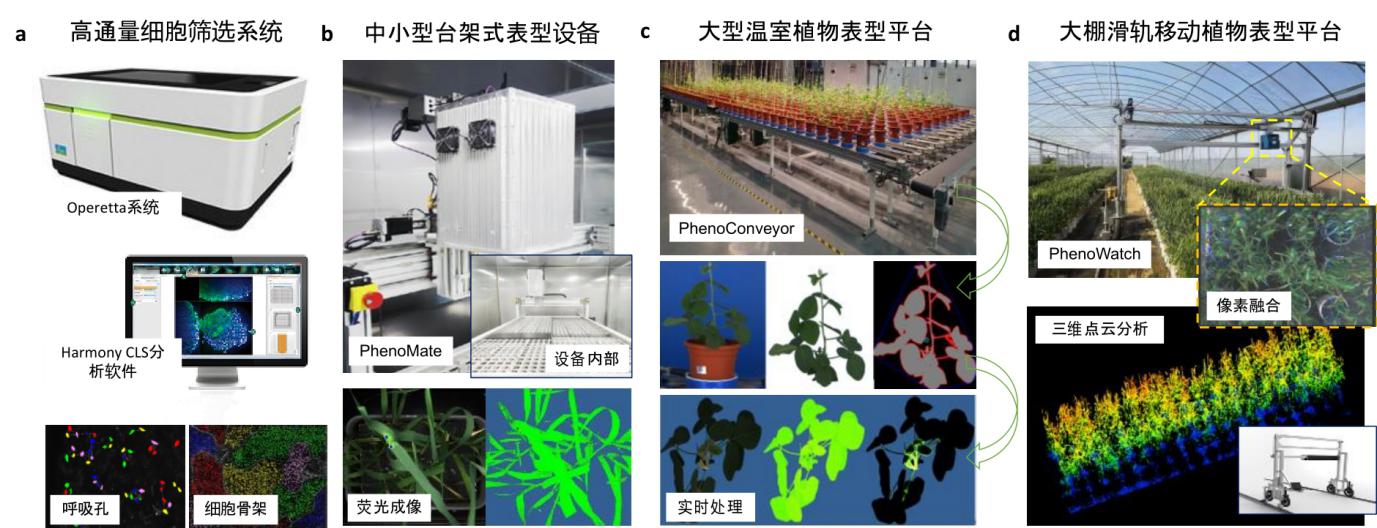
(a)高通量细胞筛选Operetta系统(图片来源: [46],[127]);(b)中小型台架式表型设备PhenoMate系统(图片来源:南京农业大学作物表型交叉研究中心);(c)大型温室植物表型平台PhenoConveyor系统(图片来源:南京农业大学作物表型交叉研究中心);(d)温室大棚中使用的滑轨移动植物表型设备PhenoWatch(图片来源:南京农业大学作物表型交叉研究中心)。
图2多尺度室内植物表型采集平台
Fig. 2 Multi-scale indoor plant phenotyping platforms
图2(a)中小型设备Operetta CLS系统可高通量追踪分析上千个植物细胞,通过旋转高速共聚焦显微镜和基于图形界面的分析软件,分析细胞和亚细胞的性状;图2(b)展示的中型的PhenoMate系统是一款对中小型植物进行顶部叶绿素荧光成像的系统,通过LED作为激发光源来激发叶绿素荧光(红色)或白色和近红外光谱,以此计算反映叶绿素含量的叶绿素指数(RChl)、花青素含量的花青素指数(RAnt)和绿素荧光和反射光谱图像;图2(c)中室内大型植物表型平台PhenoConveyor集成了多元成像装置和大量传感器,通过传送带将贴有二维码的盆栽植物传送到密闭箱体进行可见光和荧光成像,进而对植物进行多方位的性状提取(如植株顶部和侧面的面积、外接轮廓的长宽、颜色,以及光合效率等参数),平台亦可对植物生长的各个阶段进行跟踪和量化分析;图2(d)温室中使用的表型设备PhenoWatch为门架式结构,通过安装集成传感设备(包括激光雷达、多光谱和RGB成像等)于门架上,再通过固定轨道和电机驱动可将门架移动到植物上方采集图像、获取植物的高精度3D点云,以此进行小群体作物的单株识别和一系列的表型参数分析(如株高、株幅、叶长、叶倾角和叶面积等)。
4.1 大型室内植物表型平台
根据应用场景的不同,大型室内表型组学研究设施通过采集和分析可控环境条件下植物的表型信息,来实现自助化、规模化的研究目标。这类平台的表型采集方式通常可分为传送式(plant-to-sensor)和轨道式(sensor-to-plant)[128,129]。国际上大多数的室内表型设施属于传送式设计,即植物在传送带上培养,通过电动马达传送至成像区域进行拍摄;轨道式表型系统主要根据温室结构搭建可移动的采集系统,可实现植物原位成像。大型室内平台一般都配备有自动灌溉装置,可实现自动补水等实验措施。另外,盆栽都会贴有二维码以保证对植物品种、实验方式和生长情况的跟踪。传送带式表型平台能够通过增加传送带数目扩大整个室内平台的容量,同时可以根据实验设计加装定制化光传感器,因此可满足多种实验需求。然而,此类表型平台需要专业人员的长期技术支持,平台价格和维护成本较高,很难大规模推广应用。轨道式表型平台操作流程相对简单,通过一定的培训就能够掌握平台使用,而且其架设所需场地较小,也可通过改进结构、增设新型传感设备扩大监测范围。此类平台相对价格适中,其主要问题是相对有限的有效载荷限制了可集成的传感器数量,另外数据采集质量和通量略逊于全自动传送带平台。需要指出的是,大型平台一般依赖于特定的商业软件管理数据来完成表型量化分析,因此软硬件的升级、特定功能的定制和更新迭代的代价很高,需要在购置安装前特别加以注意。表1列举了一些国际上比较常见的大型室内表型平台。
表1 大型高通量室内植物表型平台介绍
Table 1 Introduction of large-scale high-throughput indoor plant phenotyping platform
| 技术方法 | 获取参数 | 平台类型 | 对应作物 | 应用方向 | |
| Scanalyzer 3D,Greenhouse Scanalyzer等 德国LemnaTec | 可见光成像、3D激光扫描、荧光成像、红外线/热成像、近红外成像、光电技术、自动控制、机电一体化技术 | 器官:叶长、叶宽、叶面积、叶角度、叶色、水分分布、茎宽、茎长、茎体积、节长、节数目、叶病斑、种子颜色、种子颜色面积 个体:结构、宽度、密度、对称性 | 大型传送带式 | 小麦、水稻、大豆、玉米、 烟草、甜瓜、豌豆等 | 植物生理学、农业科学、植物病理学、遗传育种、突变株筛选、植物形态建模、植物胁迫生理学、植物水力学等[4,55] |
| PhenoConveyor 荷兰Phenokey | 可见光成像、荧光成像,光电成像技术、自动控制、机电一体化技术。 | 个体:植株顶部和侧面,面积、轮廓宽度和长度、面积、顶部颜色等。 生理:植物暗适应后的最大光合效率成像,植物照光后的实际光合效率。 | 大型传送带式 | 小麦、大豆、水稻 | 植物形态建模、植物胁迫生理学(www.bio-equip.com/show1equip.asp?equipid=3839968) |
| HRPF系统 中国华中农业大学和华中科技大学 | 可见光成像、光电成像技术、自动控制、机电一体化技术。 | 个体:植株株高、分蘖数 器官:叶片角度、绿叶面积、鲜重、干重 | 大型传送带式 | 水稻、玉米、小麦、 油菜 | 遗传育种[5,15] |
| CropObserver 荷兰 PhenoVation | 荧光成像、自动控制和机械一体化技术 | 生理:最大光合效率、实际光合效率、光强PAR、太阳总辐射、相对电子传递速率ETR等 | 大型传送带式 | 小麦、玉米 | 群体光合作用连续监测,抗逆品种的筛选(www.phenovation.com/cropobserver) |
| WPScan,WPScanConveyor 荷兰WPS | 高光谱成像,可见光成像,多光谱成像,荧光成像,激光扫描3D成像,热成像等。 | 个体:植株反射指数、三维高光谱数据集、形态、结构、颜色指数、光合生理特征 | 大型传送带式 | 水稻、小麦、玉米、番茄、白菜 | 植物生理学、农业科学、遗传育种、植物形态建模等(www.phenotrait.com/?p=5057) |
| PhenoWatch Crop 3D 中国科学院植物研究所和上海泽泉科技股份有限公司 | 3D-LiDAR激光雷达、多光谱成像、RGB成像,可扩展热红外成像技术。 | 群体:冠层覆盖度、冠层高度 个体:株高、株幅、光谱特征、植被指数、体积 器官:叶长、叶宽、叶倾角和叶面积 | 大型门架轨道式 | 小麦、玉米、水稻 | 植物表型、作物育种、植物抗逆、生理生态、田间生产检测与管理[54] |
4.2 中小型室内植物表型设备
中小型室内表型设备主要用于监测中小型植物或者植物苗期的生长状况,一般为台式或门架式结构。此类平台的研发周期较短,需要的研发费用较低,占用场地面积小,因此很多科研机构都开发了自己特制的中小型室内表型设备。此外,这类设备能够通过定制化成像设备和机械结构来满足不同的表型采集要求,所以中小型实验单位可以针对所研究的作物选择此类平台。这类设备的一个重要研发方向是低成本、便携式和定制化,在保障表型数据获取的同时,兼顾多样化、灵活性的表型检测理念。例如奥地利科学院孟德尔研究所(Gregor Mendel Institute,GMI),维也纳生物研究中心(Vienna BioCenter,VBC)设计的开源硬件系统PhenoBox[130],根据室内实验需求开发功能独特、操作简单的箱式表型设备。虽然自动化程度低,但是价格经济、适合非专业人员使用和维护。表2列举了一部分国际上较常见的中小型室内植物表型设备。
表2 中小型室内植物表型平台介绍
Table 2 Introduction of small and medium-sized indoor plant phenotyping platform
| 技术方法 | 获取参数 | 平台类型 | 对应作物 | 应用方向 | |
| ScanalyzerHTS SeedScanalyzer 德国 LemnaTec | 可见光成像(VIS)、荧光成像、近红外成像(NIR)、多光谱成像、高光谱成像、3D激光扫描成像技术 | 个体:结构、宽度、对称性 器官:叶宽、叶面积、叶角度、叶色、叶病斑、种子颜色、种子面积 生理:植物的水分分布状态、光合荧光 | 中小型台式 | 拟南芥、稻麦等作物的早期拍摄 | 植物表型、植物生理生态,突变植株筛选、形态建模、遗传育种、植物病理、植物胁迫、种子病理、种子生理等[4,55,131-133] |
| PlantScreen 捷克Photon Systems Instruments(PSI) | 荧光成像、RGB成像、高光谱成像、热成像分析、近红外成像技术 | 个体:三维光谱数据、形态、结构 生理:叶绿素荧光、水分分布 | 中型门架式 | 玉米、水稻、小麦、 拟南芥 | 植物表型、植物生理生态,形态建模、遗传育种、植物病理、植物胁迫[8,105,134,135] |
| WIWAM Conveyor 比利时SMO,Ghent大学VIB研究室 | 荧光成像、热成像、近红外成像、高光谱成像、多光谱成像、CT断层扫描、RGB成像技术 | 个体:三维光谱数据、形态、结构。 生理:叶绿素荧光、水分分布 | 中小型台式 | 水稻、玉米、 拟南芥 | 植物表型,生理生态,形态建模、遗传育种、植物病理[136] |
| Phenoscope 法国INRA | 荧光成像 | 个体:顶部面积 | 小型台式 | 拟南芥 | 数量遗传学研究、突变筛选[137,138] |
| PhenoMate 荷兰PhenoVation;北京慧诺瑞德科技有限公司 | 荧光成像、光电技术、自动控制,机电一体化技术 | 生理:叶绿素指数、花青素指数,RGB图像 | 小型门架式 | 大豆、 拟南芥 | 基因型筛选、突变株筛选,光合作用,全叶片和植株的光合作用(www.phenovation.com/PhenoMate/)。 |
| PlantExplorer 荷兰PhenoVation;北京慧诺瑞德科技有限公司 | 多光谱叶绿素荧光、可见光成像技术 | 个体:顶部面估计 器官:种子面积 | 小型箱式 | 水稻、 烟草、柑橘、葡萄等 | 各类小型植物的生长表型(www.phenovation.com/plantexplorer/)。 |
| PhenoBox 奥地利科学院GMI研究所 | 开源植物成像技术,拍摄控制软件,开源图像数据处理 | 器官:各类叶片表型 | 小型箱式(自主研发) | 玉米、烟草 | 玉米黑穗病,叶面在胁迫下的表型变化[130] |
| HyperScanner 美国威斯康星大学麦迪逊分校 | 开源部件构建,植物胁迫实验中收集反射率曲线,可显示红外反射率谱的变化 | 器官:叶片光谱 | 小型箱式(自主研发) | 拟南芥 | 叶面在胁迫下的光谱响应[139] |
| MyROOT 2.0 西班牙拉蒙·鲁尔大学,拉萨尔大学 | 高通量根长测量设备 | 器官:根部长度,根部检测 | 小型固定式拍摄系统(自主研发) | 拟南芥 | 亚像素精度自动根部检测率[140] |
| LiDARPheno 加拿大萨斯卡通萨斯喀彻温大学 | 经济型激光雷达,悬挂式传送装置 | 器官:叶形,三维叶面积,叶色等 | 小型悬挂式(自主研发) | 兰花、室内植物 | 室内植物的三维叶片表型[141] |
4.3 室内表型数据管理和解析
表型硬件是基础,控制分析软件则是平台的灵魂。随着植物研究的不断进展,表型采集硬件(如图像传感设备)的更新换代越来越快,因此相应的控制软件、数据管理系统和性状解析算法的发展速度也日新月异。针对表型获取数据标准不统一、数据不确定性高、重复性低和时效性高等多个问题,室内表型数据管理和解析方法的重要性与日俱增。对于表型数据的标准化和管理系统可包括数据预处理、数据整合、数据存储等多方面内容。国际上通用的表型数据标准一般有ISA-Tab体系(cropnet.pl/ phenotypes)和MIAPPE体系(Minimal Information about Plant Phenotyping Experiments[142,143]。这两项标准均通过本体论来描述表型实验所需的元数据( metadata ),以此建立标准属性列表供数据管理系统使用。对于数据整合和存储系统,世界各国有着不同的解决方案,比如法国国家农业科学研究院(Institut Nationale de la Recherche Agronomigue,INRA)的PHIS(Phenotyping Hybrid Information System)系统[143]通过本体驱动架构集成和管理来自多个实验和平台的表型数据,根据实验和平台对象之间的关系完善元数据,再通过Web服务与外部数据库进行交互备份。英国诺维奇科研园(Norwich Research Park)通过物联网技术建立了CropSight小麦表型数据管理平台,通过分布式设备和云端服务器的交互完成对田间、温室和生长箱中小麦基因型的近实时监测[144]。澳大利亚莫纳什大学构建了以本体为中心结构的数据管理系统,以此捕获生物实验中的不同对象和实验条件,执行开放式的数据管理任务[145]。
对于获取表型组数据的解析,大型表型设备一般通过较为成熟的商业集成软件完成量化分析,例如著名的LemnaTec室内平台可通过LemnaGrid软件对获取的图像进行后期处理和性状分析[55,72]。同时,很多交叉研究团队也开发了一系列独立于表型采集硬件系统的表型解析软件,例如Rosette Tracker对早期拟南芥基因型的量化分析[131],Phenotiki对拟南芥形状和生长曲线的提取[146],Leaf-GP对拟南芥和小麦生长相关表型的分类[53]和RootNav 2.0通过深度学习对复杂植物根系结构的描述[147]等。对于非计算机专业的研究人员,一些成熟的图像处理应用软件也可用来完成定性的表型分析,如ImageJ、FIJI和Photoshop[148,149]。此外,对于脚本程序语言如Python或Matlab等[150,151]的使用,可以通过简单编程辅助基于图像的性状分析。这类程序的开发一般需要依赖各类图像处理软件库,如OpenCV、Scikit-Image、MATLAB Image Processing Toolbox等[150,152,153],完成图像读取、预处理、分析和性状提取。因篇幅所限,以后将单独对表型数据解析进行讨论。
5 展望和建议
植物表型组学正在进入高速发展的快车道,越来越多的植物科研人员对表型研究的方法和应用场景产生了浓厚的兴趣。中国幅员辽阔,植物种类繁多,室内植物表型平台可以针对不同生态环境中的实际研究需求,在可控条件下采集各类表型组数据。通过不断改进的传感器技术、计算机图像和人工智能等分析方法,结合各类植物研究的理论基础,为基因挖掘、遗传育种、栽培研究和农业生产管理等领域提供服务。前文对于各类室内表型设备所采集的海量数据存在的多标准、多尺度、多样化和解析方法做了简单的介绍。但是,如何有效便捷地从大数据集中提取有生物学意义的信息,进而取得新的生物学发现,现有的表型数据解析方法仍处于探索阶段。
植物表型组学是突破未来作物研究和应用的关键领域,多尺度表型数据集既可解释新的生物学现象,也可为育种、栽培和农业生产实践提供大数据支持。特别是在可控的室内条件下,表型数据的采集首先需要考虑到多尺度数据集的校准和融合。单个传感器获取的数据非常有限,但是多种传感器则存在制式标准和同步校准的技术问题。结合多种传感器或成像技术的确可以获得更全面的表型特征,但提高数据的可信度,则需要结合传统方法和多源数据的交叉验证来完成。同时,融合不同的成像技术也可突破单一成像技术的局限,例如叶片的生长发育可通过可见光成像得到叶片的大小和颜色等参数,在结合多光谱和高光谱技术后可测算叶片的营养成分和微量元素含量,加载激光雷达或CT成像后还可获取叶片卷曲度和三维叶面积等空间结构信息,再通过显微成像可对细胞层面的表型(如气孔、叶脉等)完成获取,最终对叶片生长发育提供从细胞到器官的一整套表型组数据。然而上述技术的综合应用和多源数据融合对于世界各国的表型研究团队仍是一大技术难题。
最新的人工智能植物生长室、植物工厂和快速迭代育种(speed breeding)也为室内表型平台技术提供了新的发展方向[49,154]。通过精准控制室内的温湿度、光照度、气体浓度、风量风速等参数,可以为封闭空间内的植物科学研究提供精确的气候环境模拟,以此完成深度调控下的作物全生育期生长表型参数采集与分析。表型组学与其它组学(如基因组学、转录组学、蛋白组学和代谢组学等)的结合将更全面地绘制出植物生长发育过程中的调控网络和功能基因表达,最终为揭示植物的生物学规律和加速作物研究的进程提供必要的研究平台。随着室内表型平台集成度的不断提升,表型性状数据的获取也将愈发全面。但是如何将室内表型技术与生物实验无缝结合,推动遗传学、作物学和农业生产的快速发展,这需要植物表型研究团队和生物研究团队的通力合作。针对重要表型性状,设计跨学科实验,通过溯源跟踪植物的生长,获得可靠的全生育期的表型组数据。同时,针对不同植物的研究需求建立不同的分析标准和流程,保障表型研究的可靠性和延续性,进而为建立全国共享的植物表型平台系统服务。以此为契机,加速我国各级研究机构的成果交流,减少重复研发和重复投资。
针对中国目前室内表型平台装置主要依赖进口、依靠国外商业机构研发的现状,中国表型研究团队可以考虑与国内制造企业合作。在提高原创性的基础上,有选择地启动产学一体的研发计划。中国自动化和光电技术水平在世界上举足轻重,通过与相关领域的专业融合、协同研发,才能研发出适合中国的表型设备和相应的关键技术。表型分析需要多学科协同合作,在优化表型数据存储结构、制定通行全国的表型组数据标准的基础上,系统提取“细胞-组织-器官-个体-群体”等多层次性状,有针对性的挖掘还未为人所知的产量、品质和抗性的综合表型性状。
在不久的将来,如何构建自动化集成表型采集平台获得多尺度、多条件及多物种的海量表型组数据,如何融合海量表型数据集、建立技术体系保障表型组数据的质量,如何建立分布式(多地点)和集中式(云端)互补的关键采集分析技术等问题,将伴随着植物表型组学研究的不断深入细化。在保障数据质量和完整性后,从表型组数据中动态解析表型性状,结合数据挖掘、计算机图像、生物信息学、数理统计和机器学习等方法,通过多学科交叉解答作物研究中的实际问题也将成为表型研究的重要方向之一。然而以上的各项任务均需要优秀复合型人才的引领和参与,需要不同学科背景、开放性创新团队的协作。人才资源是保障综合国力的最重要的战略资源,因此需要我们改变固有的人才培养思路,在植物研究中鼓励交叉创新人才的快速成长。不盲目跟踪模仿其他国家技术,建设我们自己学科交叉、优势互补、紧密合作的新型科研团队。同时在全国范围内构建知识和人才的共享平台,将会是中国植物表型研究能否引领世界的关键。
参考文献
[1] Araus J L, Cairns J E. Field high-throughput phenotyping: the new crop breeding frontier[J]. Trends Plant Sci, 2014, 19(1): 52-61.
[2] Crain J, Mondal S, Rutkoski J, et al. Combining high-throughput phenotyping and genomic information to increase prediction and selection accuracy in wheat breeding[J]. Plant Genome, 2018, 11: no.1.
[3] Tester M, Langridge P. Breeding technologies to increase crop production in a changing world[J]. Science, 2010, 327(5967): 818-822.
[4] Chen D, Neumann K, Friedel S, et al. Dissecting the phenotypic components of crop plant growth and drought responses based on high-throughput image analysis[J]. Plant Cell Online, 2014, 26(12): 4636-4655.
[5] Yang W, Guo Z, Huang C, et al. Combining high-throughput phenotyping and genome-wide association studies to reveal natural genetic variation in rice[J]. Nat Commun, 2014, 5: no.5087.
[6] Houle D, Govindaraju D R, Omholt S. Phenomics: the next challenge[J]. Nat Rev Genet, 2010, 11(12): 855-866.
[7] White J W, Andrade-Sanchez P, Gore M A, et al. Field-based phenomics for plant genetics research[J]. F Crop Res, 2010, 133: 101-112.
[8] Junker A, Muraya M M, Weigelt-Fischer K, et al. Optimizing experimental procedures for quantitative evaluation of crop plant performance in high throughput phenotyping systems[J]. Front Plant Sci, 2015, 5: no.770.
[9] Nguyen G N, Kant S. Improving nitrogen use efficiency in plants: Effective phenotyping in conjunction with agronomic and genetic approaches[J]. Funct Plant Biol, 2018, 45(6): 606-619.
[10] Cendrero-Mateo M P, Muller O, Albrecht H, et al. Field phenotyping: Concepts and examples to quantify dynamic plant traits across scales in the field[J]. Terrestrial Ecosystem Research Infrastructures: Challenges and Opportunities, Boca Raton: CRC Press,2017: 53-80.
[11] Zhao C, Zhang Y, Du J. Crop phenomics: Current status and perspectives[J]. Front Plant Sci, 2019, 10: no.714.
[12] Furbank R T, Tester M, Berry S, et al. Phenomics–technologies to relieve the phenotyping bottleneck[J]. Trends Plant Sci, 2011, 16(12): 635-644.
[13] Tardieu F, Cabrera-Bosquet L, Pridmore T, et al. Plant phenomics, from sensors to knowledge[J]. Curr Biol, 2017, 27(15): 770-783.
[14] Puijalon S, Bouma T J, Douady C J, et al. Plant resistance to mechanical stress : Evidence of an avoidance-tolerance trade-off[J], New Phytologist, 2011, 191(4): 1141-1149.
[15] Yang W, Feng H, Zhang X, et al. Crop phenomics and high-throughput phenotyping: Past decades, current challenges, and future perspectives[J]. Mol Plant, 2020, 13: 187-214.
[16] Kirchgessner N, Liebisch F, Yu K, et al. The ETH field phenotyping platform FIP: A cable-suspended multi-sensor system[J]. Funct Plant Biol, 2017, 44(1): 154-168.
[17] Roy J, Tardieu F, Tixier-Boichard M, et al. European infrastructures for sustainable agriculture[J]. Nat Plants, 2017, 3: 756-758
[18] Pieruschka R, Schurr U. Plant phenotyping: Past, present, and future[J]. Plant Phenomics, 2019: no. 7507131.
[19] Rosenqvist E, Großkinsky D K, Ottosen C O, et al. The phenotyping dilemma-the challenges of a persified phenotyping community[J]. Front Plant Sci, 2019, 10: no.163.
[20] Cobb J N, DeClerck G, Greenberg A, et al. Next-generation phenotyping: Requirements and strategies for enhancing our understanding of genotype-phenotype relationships and its relevance to crop improvement[J]. Theor Appl Genet, 2013, 126(4): 867-887.
[21] Tardieu F, Simonneau T, Muller B. The Physiological Basis of Drought Tolerance in Crop Plants: A Scenario-Dependent Probabilistic Approach[J]. Annu Rev Plant Biol, 2018, 69(1), 733-759.
[22] 周济, Tardieu F, Pridmore T P, 等. 植物表型组学:发展、现状与挑战[J]. 南京农业大学学报, 2018, 41(4): 580-588.
Zhou J, Tardieu F, Pridmore T P, et al. Plant phenomics: History, present status and challenges[J]. J Nanjing Agric Univ, 2018, 41(4): 580-588.
[23] Reynolds D, Baret F, Welcker C, et al. What is cost-efficient phenotyping? Optimizing costs for different scenarios[J]. Plant Sci, 2019, 282: 14-22.
[24] 高宇, 高军萍, 李寒, 等. 植物表型监测技术研究进展及发展对策[J]. 江苏农业科学, 2017, 45(11): 5-10.
Gao Y, Gao J, Li H, et al. Research progress and development countermeasure of plant phenotypic monitoring technology[J]. Jiangsu Agricultural Sciences, 2017, 45(11): 5-10.
[25] 郭庆华, 杨维才, 吴芳芳, 等. 高通量作物表型监测:育种和精准农业发展的加速器[J]. 中国科学院院刊, 2018, 33(9): 940-946.
Guo Q, Yang W, Wu F, et al. High-throughput crop phenotyping: Accelerators for development of breeding and precision agriculture[J]. Bulletin of Chinese Academy of Science, 2018, 33(9): 940-946.
[26] 程超华, 唐蜻, 邓灿辉, 等. 表型组学及多组学联合分析在植物种质资源精准鉴定中的应用[J/OL]. 分子植物育种.http://kns.cnki.net/kcms/detail/46.1068.s.20190517.1331.002.html
Cheng C, Tang Q, Deng C, et al. Application of phenomics and multiomics joint analysis in accurate identification of plant germplasm resources[J/OL]. Molecular Plant Breeding.http://kns.cnki.net/kcms/detail/46.1068.s.20190517.1331.002.html.
[27] 刘立才, 梁丽秀, 杜传红. 植物表型组学在现代农业中的应用[J]. 科技创新与应用, 2019, (22): 169-174.
[28] 杨贵军, 李长春, 于海洋, 等. 农用无人机多传感器遥感辅助小麦育种信息获取[J]. 农业工程学报, 2015, 31(21): 184-190.
Yang G, Liang L, Yu H, et al. UAV based multi-load remote sensing technologies for wheat breeding information acquirement[J]. Transaction of the CSAE, 31(21): 184-190.
[29] 刘建刚, 赵春江, 杨贵军, 等. 无人机遥感解析田间作物表型信息研究进展[J]. 农业工程学报, 2016, 32(24): 98-106.
Liu J, Zhao C, Yang G, et al. Review of field-based phenotyping by unmanned aerial vehicle remote sensing platform[J]. Transaction of the CSAE, 2016, 32(24): 98-106.
[30] 高林, 杨贵军, 李红军, 等. 基于无人机数码影像的冬小麦叶面积指数探测研究[J]. 中国生态农业学报 2016, 24(9): 1254-1264.
Gao L, Yang G, Li H, et al. Winter wheat LAI estimation using unmanned aerial vehicle RGB-imaging[J]. Chinese Agricultural Science Bulletin, 2016, 24(9): 1254-1264.
[31] 温维亮, 郭新宇, 赵春江, 等. 基于三维数字化的玉米株型参数提取方法研究[J]. 中国农业科学, 2018, 51(6): 1034-1044.
Wen W, Guo X, Zhao C, et al. Research on maize plant type parameter extraction by using three dimensional digitizing data[J]. Scientia Agricultura Sinica, 2018, 51(6): 1034-1044.
[32] 曹卫星, 朱艳, 田永超, 等. 作物精确栽培技术的构建与实现[J]. 中国农业科学, 2011, 44(19): 3955-3969.
Cao W, Zhu Y, Tian Y, et al. Development and Implementation of Crop precision cultivation technology[J]. Scientia Agricultura Sinica, 2011, 44(19): 3955-3969.
[33] 马孟莉, 朱艳, 李文龙, 等. 基于分层多端元混合像元分解的水稻面积信息提取[J]. 农业工程学报, 2012, 28(2): 154-159.
Ma M, Zhu Y, Li W, et al. Extracting area information of paddy rice based on stratified multiple endmember spectral mixture analysis[J]. Transactions of the CSAE, 2012, 28(2): 154-159.
[34] 郭小清, 胥晓明, 曹卫星, 等. 作物模型系统 Web 服务集成方法[J]. 农业工程学报, 2013, 29(22): 162-170.
Guo X, Xu X, Cao W, et al. Web service integration method of crop model system[J]. Transactions of the CSAE, 2013, 29(22): 162-170.
[35] 胡伟娟, 凌宏清, 傅向东. 植物表型组学研究平台建设及技术应用[J]. 遗传, 2019, 41(11): 1060-1066.
Hu W, Ling H, Fu X. Development and application of the plant phenomics analysis platform[J]. Hereditas(Beijing), 2019, 41(11): 1060-1066.
[36] 赵春江. 植物表型组学大数据及其研究进展[J]. 农业大数据学报, 2019, 1(2): 5-14.
Zhao C. Big data of plant phenomics and its research progress[J]. Journal of Agricultural Big Data, 2019, 1(2): 5-14.
[37] 唐惠燕, 倪峰, 李小涛, 等. 基于Scopus的植物表型组学研究进展分析[J]. 南京农业大学学报, 2018, 41(6): 1133-1141.
Tang H, Ni F, Li X. Analysis of the advance in plant phenomics research based on Scopus tools[J]. Journal of Nanjing Agricultural University, 2018, 41(6): 1133-1141.
[38] 李晓曼, 张扬, 徐倩. 基于文献计量的植物表型组学研究进展分析[J]. 农业大数据学报, 2019, 1(2): 64-75
Li X, Zhang Y, Xu Q, et al. Bibliometrics-based analysis of advances in plant phenomics research[J]. Journal of Agricultural Big Data, 2019, 1(2): 64-75.
[39] 段凌凤, 杨万能. 水稻表型组学研究概况和展望[J]. 生命科学, 2016 28(10): 1129-1137.
Duan L, Yang W. Research advances and future scenarios of rice phenomics[J]. Chinese Bulletin of Life Sciences, 2016, 28(10): 1129-1137.
[40] 胡伟娟, 傅向东, 陈凡, 等. 新一代植物表型组学的发展之路[J]. 植物学报, 2019, 54(5): 558-568.
Hu W, Fu X, Chen F, et al. A path to next generation of plant phenomics[J]. Bulletin of Botany, 2019, 54(5): 558-568.
[41] 潘映红. 论植物表型组和植物表型组学的概念与范畴[J]. 作物学报, 2015, 41(2): 175-186.
Pan Y. Analysis of concepts and categories of plant phenome and phenomics[J]. Acta Agronomica Sinica, 41(2): 175-186.
[42] Fiorani F, Schurr U. Future scenarios for plant phenotyping[J]. Annu Rev Plant Biol, 2013, 64(1): 267-291.
[43] Gibbs J A, Pound M, French A P, et al. Approaches to three-dimensional reconstruction of plant shoot topology and geometry[J]. Funct Plant Biol, 2017, 44: 62-75.
[44] Beck M, Zhou J, Faulkner C, et al. Spatio-temporal cellular dynamics of the Arabidopsis flagellin receptor reveal activation status-dependent endosomal sorting[J]. Plant Cell, 2012, 24(10): 4205-4219.
[45] Zhou J, Spallek T, Faulkner C, et al. CalloseMeasurer: A novel software solution to measure callose deposition and recognise spreading callose patterns[J]. Plant Methods, 2013, 8: no. 49.
[46] Faulkner C, Zhou J, Evrard A, et al. An automated quantitative image analysis tool for the identification of microtubule patterns in plants[J]. Traffic, 2017, 18(10): 683-693.
[47] Singh V, Misra A K. Detection of plant leaf diseases using image segmentation and soft computing techniques[J]. Inf Process Agric, 2017, 4(1): 41-49.
[48] Zhou J, Reynolds D, Websdale D, et al. CropQuant: An automated and scalable field phenotyping platform for crop monitoring and trait measurements to facilitate breeding and digital agriculture[J]. bioRxiv, 2017. https://doi.org/10.1101/161547.
[49] Watson A, Ghosh S, Williams M J, et al. Speed breeding: A powerful tool to accelerate crop research and breeding[J]. Nat Plants, 2018, 4: 23-29.
[50] Cao Z, Yao X, Liu H, et al. Comparison of the abilities of vegetation indices and photosynthetic parameters to detect heat stress in wheat[J]. Agric For Meteorol, 2019, 265: 121-136.
[51] Santos T T, Koenigkan L V, Barbedo J G A, et al. 3d plant modeling: Localization, mapping and segmentation for plant phenotyping using a single hand-held camera[C]// Lect Notes Comput Sci (including Subser Lect Notes Artif Intell Lect Notes Bioinformatics), 2015, 8928: 247-263.
[52] Chen D, Shi R, Pape J M, et al. Predicting plant biomass accumulation from image-derived parameters[J]. Gigascience, 2018, 7(2): 1-13.
[53] Zhou J, Applegate C, Alonso A D, et al. Leaf-GP: An open and automated software application for measuring growth phenotypes for arabidopsis and wheat[J]. Plant Methods, 2017, 13(1): 1-30.
[54] Guo Q, Wu F, Pang S, et al. Crop 3D-a LiDAR based platform for 3D high-throughput crop phenotyping[J]. Sci China Life Sci, 2018, 61(3): 328-339.
[55] Arvidsson S, Pérez-Rodríguez P, Mueller-Roeber B. A growth phenotyping pipeline for Arabidopsis thaliana integrating image analysis and rosette area modeling for robust quantification of genotype effects[J]. New Phytol, 2011, 191(3): 895-907.
[56] Sadras V O, Slafer G A. Environmental modulation of yield components in cereals: Heritabilities reveal a hierarchy of phenotypic plasticities[J]. F Crop Res, 2012, 127: 215-224.
[57] Zhu J, van der Werf W, Anten N P R, et al. The contribution of phenotypic plasticity to complementary light capture in plant mixtures[J]. New Phytol, 2015, 207(4): 1213-1222.
[58] Huang X, Wei X, Sang T, et al. Genome-wide association studies of 14 agronomic traits in rice landraces[J]. Nat Genet, 2010, 42(11): 961-967.
[59] Yang W, Guo Z, Huang C, et al. Genome-wide association study of rice (Oryza sativa L.) leaf traits with a high-throughput leaf scorer[J]. J Exp Bot, 2015, 66(18): 5605-5615.
[60] Duan T, Chapman S C, Holland E, et al. Dynamic quantification of canopy structure to characterize early plant vigour in wheat genotypes[J]. J Exp Bot, 2016, 67: 4523-4534.
[61] Scheres B, van der Putten W H. The plant perceptron connects environment to development[J]. Nature, 2017, 543(7645): 337-345.
[62] Reuzeau C, Pen J, Frankard V, et al. TraitMill: A discovery engine for identifying yield-enhancement genes in cereals[J]. Plant Gene Trait, 2010, 3(1): 1-6.
[63] Palosuo T, Kersebaum K C, Angulo C, et al. Simulation of winter wheat yield and its variability in different climates of Europe: A comparison of eight crop growth models[J]. Eur J Agron, 2010, 35(3): 103-114.
[64] Bulman P, Hunt L A. Relationships among tillering, spike number and grain yield in winter wheat (Triticum aestivum L.) in Ontario[J]. Can J Plant Sci, 1988, 68(3): 583-596.
[65] Wu X, Chang X, Jing R. Genetic insight into yield-associated traits of wheat grown in multiple rain-fed environments[J]. PLoS One, 2012, 7(2): no.e31249.
[66] Topp C N, Iyer-Pascuzzi A S, Anderson J T, et al. 3D phenotyping and quantitative trait locus mapping identify core regions of the rice genome controlling root architecture[J]. Proc Natl Acad Sci U S A, 2013, 110(18): 1695-1704.
[67] Xie Q, Fernando K M C C, Mayes S, et al. Identifying seedling root architectural traits associated with yield and yield components in wheat[J]. Ann Bot, 2017, 119(7): 1115-1129.
[68] Wu J, Wu Q, Pagès L, et al. RhizoChamber-Monitor: A robotic platform and software enabling characterization of root growth[J]. Plant Methods, 2018, 14(1): 1-15.
[69] Zhang C, Si Y, Lamkey J, et al. High-throughput phenotyping of seed/seedling evaluation using digital image analysis[J]. Agronomy, 2018, 8(5): 1-14.
[70] Alkhudaydi T, Reynolds D, Griffiths S, et al. An exploration of deep-learning based phenotypic analysis to detect spike regions in field conditions for UK bread wheat[J]. Plant Phenomics, 2019, 1: 1-17.
[71] Schmidt J, Claussen J, Wörlein N, et al. Drought and heat stress tolerance screening in wheat using computed tomography[J]. Plant Methods, 2020, 16: 1-12.
[72] Campbell M T, Knecht A C, Berger B, et al. Integrating image-based phenomics and association analysis to dissect the genetic architecture of temporal salinity responses in rice[J]. Plant Physiol, 2015, 168(4): 1476-1489.
[73] Cabrera-Bosquet L, Fournier C, Brichet N, et al. High-throughput estimation of incident light, light interception and radiation-use efficiency of thousands of plants in a phenotyping platform[J]. New Phytol, 2016, 212(1): 269-281.
[74] Nguyen G N, Maharjan P, Maphosa L, et al. A robust automated image-based phenotyping method for rapid vegetative screening of wheat germplasm for nitrogen use efficiency[J]. Front Plant Sci, 2019, 10: 1-15.
[75] Jahnke S, Roussel J, Hombach T, et al. Pheno seeder - A robot system for automated handling and phenotyping of inpidual seeds[J]. Plant Physiol, 2016, 172: 1358-1370.
[76] Liang X, Wang K, Huang C, et al. A high-throughput maize kernel traits scorer based on line-scan imaging[J]. Meas J Int Meas Confed, 2016, 90: 453-460.
[77] Shao M, Zhang Y, Du J, et al. Fast analysis of maize kernel plumpness characteristics through Micro-CT technology[C]// Int. Conf. Comput. Comput. Technol. Agric. , 2017: 31-39.
[78] Rahman A, Cho B K. Assessment of seed quality using non-destructive measurement techniques: A review[J]. Seed Sci Res, 2016, 26: 285-305.
[79] Caporaso N, Whitworth M B B, Fisk I D D. Near-Infrared spectroscopy and hyperspectral imaging for non-destructive quality assessment of cereal grains[J]. Appl Spectrosc Rev, 2018, 53: 667-687.
[80] Nakamura S, Satoh H, Ohtsubo K N I. Development of formulae for estimating amylose content, amylopectin chain length distribution, and resistant starch content based on the iodine absorption curve of rice starch[J]. Biosci Biotechnol Biochem, 2015, 79(3): 443-455.
[81] Herzig P, Backhaus A, Seiffert U, et al. Genetic dissection of grain elements predicted by hyperspectral imaging associated with yield-related traits in a wild barley NAM population[J]. Plant Sci, 2019, 285: 151-164.
[82] Crowell S, Falcao A X, Shah A, et al. High-resolution inflorescence phenotyping using a novel image-analysis pipeline, PANorama[J]. Plant Physiol, 2014, 165(2): 479-495.
[83] Shewry P R. Wheat[J]. J Exp Bot, 2019, 60: 1537-1553.
[84] Sacco A, Di Matteo A, Lombardi N, et al. Quantitative trait loci pyramiding for fruit quality traits in tomato[J]. Mol Breed, 2013, 31(1): 217-222.
[85] Folta K M, Carvalho S D. Photoreceptors and control of horticultural plant traits[J]. HortScience, 2015, 50: 1274-1280.
[86] Zhu G, Wang S, Huang Z, et al. Rewiring of the fruit metabolome in tomato breeding[J]. Cell, 2018, 172(1/2): 249-261.
[87] Fang C, Luo J. Metabolic GWAS-based dissection of genetic bases underlying the persity of plant metabolism[J]. Plant J, 2019, 97(1): 91-100.
[88] Candido V, Miccolis V, Rivelli A R. Yield traits and water and nitrogen use efficiencies of bell pepper grown in plastic-greenhouse[J]. Ital J Agron, 2009 4(3): 91-100.
[89] Kibinza S, Bazin J, Bailly C, et al. Catalase is a key enzyme in seed recovery from ageing during priming[J]. Plant Sci, 2011, 181(3): 309-315.
[90] FAO. The impact of disasters and crises on agriculture and Food Security [EB/OL]. [2020-3-10]. https://www.preventionweb.net/publications/view/57488.
[91] Touchette B W, Smith G A, Rhodes K L, et al. Tolerance and avoidance: Two contrasting physiological responses to salt stress in mature marsh halophytes Juncus roemerianus Scheele and Spartina alterniflora Loisel[J]. J Exp Mar Bio Ecol, 2009, 380(1/2): 106-112.
[92] Rausher M D. Co-evolution and plant resistance to natural enemies[J]. Nature, 2011, 411(6839): 857-864.
[93] Treutter D. Significance of flavonoids in plant resistance and enhancement of their biosynthesis[J]. Plant Biol, 2005, 7(6): 581-591.
[94] Treutter D. Significance of flavonoids in plant resistance: A review[J]. Environ Chem Lett, 2006, 4(3): 147-157.
[95] 屈宝香. 从粮食生产周期变化看中国粮食安全[J]. 作物杂志, 2004, (1): 6-9.
[96] 何斌, 武建军,吕爱锋. 农业干旱风险研究进展[J]. 地理科学进展, 2010, 29(5): 557-564.
He B, Wu J, Lv A. New advances in agricultural drought risk study[J]. Progress in Geography, 2010, 29(5): 557-564.
[97] Guo Z, Yang W, Chang Y, et al. Genome-wide association studies of image traits reveal genetic architecture of drought resistance in rice[J]. Mol Plant, 2018, 11(6): 789-805.
[98] Xiong L, Wang R, Mao G, et al. Identification of drought tolerance determinants by genetic analysis of root response to drought stress and abscisic acid[J]. Plant Physiol, 2006, 142(3): 1065-1074.
[99] Bao Y, Aggarwal P, Robbins N E, et al. Plant roots use a patterning mechanism to position lateral root branches toward available water[J]. Proc Natl Acad Sci U S A, 2014, 111(25): 9319-9324.
[100] Shiono K, Hashizaki R, Nakanishi T, et al. Multi-imaging of cytokinin and abscisic acid on the roots of rice (Oryza sativa) using matrix-assisted laser desorption/ionization mass spectrometry[J]. J Agric Food Chem, 2017, 65(35): 7624-7628.
[101] Pflugfelder D, Metzner R, Dusschoten D, et al. Non-invasive imaging of plant roots in different soils using magnetic resonance imaging (MRI)[J]. Plant Methods, 2017, 13(1): 1-9.
[102] Atkinson J A, Pound M P, Bennett M J, et al. Uncovering the hidden half of plants using new advances in root phenotyping[J]. Curr Opin Biotechnol, 2019, 55: 1-8.
[103] Karwa S, Bahuguna R N, Chaturvedi A K, et al. Phenotyping and characterization of heat stress tolerance at reproductive stage in rice (Oryza sativa L.)[J]. Acta Physiol Plant, 2020, 29: no.42.
[104] Marko D, El-Shershaby A, Carriero F, et al. Identification and characterization of a thermotolerant TILLING allele of heat shock binding protein 1 in tomato. Genes (Basel)[J]. 2019, 10: no.516.
[105] Humplík J F, Lazár D, Fürst T, et al Automated integrative high-throughput phenotyping of plant shoots: a case study of the cold-tolerance of pea (Pisum sativum L.)[J]. Plant Methods, 2015, 11(1): 1-11.
[106] Mishra K B, Mishra A, Kubásek J, et al. Low temperature induced modulation of photosynthetic induction in non-acclimated and cold-acclimated Arabidopsis thaliana: chlorophyll a fluorescence and gas-exchange measurements[J]. Photosynth Res, 2019, 139(1-3): 123-143.
[107] Moura D S, Brito G G, Moraes Í L, et al. Cold tolerance in rice plants: Phenotyping procedures for physiological breeding[J]. J Agric Sci, 2017, 10(1): 313-324.
[108] Deinlein U, Stephan A B, Horie T, et al. Plant salt-tolerance mechanisms[J]. Trends Plant Sci, 2014, 19(6): 371-379.
[109] Mehta P, Jajoo A, Mathur S, et al. Chlorophyll a fluorescence study revealing effects of high salt stress on Photosystem II in wheat leaves[J]. Plant Physiol Biochem, 2010, 48(1): 16-20.
[110] Asif M A, Schilling R K, Tilbrook J, et al. Mapping of novel salt tolerance QTL in an Excalibur × Kukri doubled haploid wheat population[J]. Theor Appl Genet, 2018, 131: 2179-2196.
[111] Bochicchio R, Sofo A, Terzano R, et al. Root architecture and morphometric analysis of Arabidopsis thaliana grown in Cd/Cu/Zn-gradient agar dishes: A new screening technique for studying plant response to metals[J]. Plant Physiol Biochem, 2015, 91: 20-27
[112] Kopittke P M, Punshon T, Paterson D J, et al. Synchrotron-Based X-Ray Fluorescence Microscopy as a Technique for Imaging of Elements in Plants[J]. Plant Physiol, 2018, 178: 507-523.
[113] Rodrigues E S, Gomes M H F, Duran N M, et al. Laboratory microprobe X-ray fluorescence in plant science: Emerging applications and case studies[J]. Front Plant Sci, 2018, 871: 1-15.
[114] Kopittke P M, de Jonge M D, Wang P, et al. Laterally resolved speciation of arsenic in roots of wheat and rice using fluorescence-XANES imaging[J]. New Phytol, 2014, 201: 1251-1262.
[115] Draz I S, Abou-Elseoud M S, Kamara A E M, et al. Screening of wheat genotypes for leaf rust resistance along with grain yield[J]. Ann Agric Sci, 2015, 60(1): 29-39.
[116] Oliva R, Ji C, Atienza-Grande G, et al. Broad-spectrum resistance to bacterial blight in rice using genome editing[J]. Nat Biotechnol, 2019, 37: 1344-1350.
[117] Kuska M, Wahabzada M, Leucker M, et al. Hyperspectral phenotyping on the microscopic scale: Towards automated characterization of plant-pathogen interactions[J]. Plant Methods, 2015, 11(1): no. 28.
[118] Omran E S E. Early sensing of peanut leaf spot using spectroscopy and thermal imaging[J]. Arch Agron Soil Sci, 2017, 63: 883-896.
[119] Singh A K, Ganapathysubramanian B, Sarkar S, et al. Deep learning for plant stress phenotyping : Trends and future perspectives machine learning in plant science[J]. Trends Plant Sci, 2018, 23(10): 883-898.
[120] Rothwell C T, Singh D, van Ogtrop F, et al. Rapid phenotyping of adult plant resistance in barley (Hordeum vulgare) to leaf rust under controlled conditions[J]. Plant Breed, 2019, 138: 51-61.
[121] Jones J D G, Dangl J L. The plant immune system[J]. Nature, 2006, 444: 323-329.
[122] Thoen M P M, Kloth K J, Wiegers G L, et al. Automated video tracking of thrips behavior to assess host-plant resistance in multiple parallel two-choice setups[J]. Plant Methods, 2016, 12: 1-12.
[123] El-Maarouf-Bouteau H, Mazuy C, Corbineau F, et al. DNA alteration and programmed cell death during ageing of sunflower seed[J]. J Exp Bot, 2011, 62(14): 5003-5011.
[124] Hu D, Ma G, Wang Q, et al. Spatial and temporal nature of reactive oxygen species production and programmed cell death in elm (Ulmus pumila L.) seeds during controlled deterioration[J]. Plant Cell Environ, 2012, 35(11): 2045-2059.
[125] Beck M, Zhou J, Faulkner C, et al. High-throughput imaging of plant immune responses[J]. Plant-Pathogen Interactions, 2014, 27(11): 67-80.
[126] Minina E A, Smertenko A P, Bozhkov P V, Metacaspase releases the brakes on autophagy[J]. Autophagy, 2014, 10(5): 926-927.
[127] Bourdais G, McLachlan D H, Rickett L M, et al. The use of quantitative imaging to investigate regulators of membrane trafficking in Arabidopsis stomatal closure[J]. Traffic, 2019, 20: 168-180.
[128] Li L, Zhang Q, Huang D. A review of imaging techniques for plant phenotyping[J]. Sensors (Switzerland), 2014, 14(11): 20078-20111.
[129] Roitsch T, Cabrera-Bosquet L, Fournier A, et al. Review: New sensors and data-driven approaches-A path to next generation phenomics[J]. Plant Sci, 2019, 282: 2-10.
[130] Czedik-Eysenberg A, Seitner S, Güldener U, et al. The ‘PhenoBox’, a flexible, automated, open-source plant phenotyping solution[J]. New Phytol, 2018, 219(2): 808-823.
[131] de Vylder J, Vandenbussche F, Hu Y, et al. Rosette Tracker: An open source image analysis tool for automatic quantification of genotype effects[J]. Plant Physiol, 2012, 160(3): 1149-1159.
[132] Omidbakhshfard M A, Fujikura U, Olas J J, et al. GROWTH-REGULATING FACTOR 9 negatively regulates arabidopsis leaf growth by controlling ORG3 and restricting cell proliferation in leaf primordia[J]. PLOS Genetics, 2018, 14(7): no.e1007484.
[133] Babst B A, Gao F, Acosta-Gamboa L M, et al. Three NPF genes in Arabidopsis are necessary for normal nitrogen cycling under low nitrogen stress[J]. Plant Physiol Biochem, 2019, 143: 1-10.
[134] Marchetti C F, Ugena L, Humplík J F, et al. A novel image-based screening method to study water-deficit response and recovery of barley populations using canopy dynamics phenotyping and simple metabolite profiling[J]. Front Plant Sci, 2019, 10(10): 1-20.
[135] Paul K, Sorrentino M, Lucini L, et al. Understanding the biostimulant action of vegetal-derived protein hydrolysates by high-throughput plant phenotyping and metabolomics: A case study on tomato[J]. Front Plant Sci, 2019, 10: no. 47.
[136] Dhondt S, van Haerenborgh D, van Cauwenbergh C, et al. Quantitative analysis of venation patterns of Arabidopsis leaves by supervised image analysis[J]. Plant J, 2012, 69(3): 553-563.
[137] Tisné S, Serrand Y, Bach L, et al. Phenoscope: An automated large-scale phenotyping platform offering high spatial homogeneity[J]. Plant J, 2013, 74(3): 534-544.
[138] Viaud G, Loudet O, Cournède P H. Leaf segmentation and tracking in Arabidopsis thaliana combined to an organ-scale plant model for genotypic differentiation[J]. Front Plant Sci, 2017, 7: no.2057.
[139] Lien M R, Barker R J, Ye Z, et al. A low-cost and open-source platform for automated imaging[J]. Plant Methods, 2019, 15(1): 1-14.
[140] González A, Sevillano X, Betegón-Putze I, et al. MyROOT 2.0: An automatic tool for high throughput and accurate primary root length measurement[J]. Comput Electron Agric, 2020, 168: no.105125.
[141] Panjvani K, Dinh A V, Wahid K A. LiDARPheno -A low-cost LiDAR-based 3D scanning system for leaf morphological trait extraction[J]. Front Plant Sci, 2019, 10: no.147.
[142] Ćwiek-Kupczyńska H, Altmann T, Arend D, et al. Measures for interoperability of phenotypic data: minimum information requirements and formatting[J]. Plant Methods, 2016, 12(1): no. 44.
[143] Neveu P, Tireau A, Hilgert N, et al. Methods dealing with multi-source and multi-scale information in plant phenomics : The ontology-driven Phenotyping hybrid Information System[J]. New Phytol, 2019, 221(1): 588-601.
[144] Reynolds D, Ball J, Bauer A, et al. CropSight: A scalable and open-source information management system for distributed plant phenotyping and IoT-based crop management[J]. Gigascience, 2019, 8(3): 1-11.
[145] Li Y, Kennedy G, Ngoran F, et al. An ontology-centric architecture for extensible scientific data management systems[J]. Futur Gener Comput Syst, 2013, 29(2): 641-653.
[146] Minervini M, Giuffrida M V., Perata P, et al. Phenotiki: An open software and hardware platform for affordable and easy image-based phenotyping of rosette-shaped plants[J]. Plant J, 2017, 90(1): 204-216.
[147] Yasrab R, Atkinson J A, Wells D M, et al. RootNav 2.0: Deep learning for automatic navigation of complex plant root architectures[J]. Gigascience, 2019, 8(11): 1-16.
[148] Collins T. ImageJ for microscopy[J]. Biotechniques, 2007, 43(Supp1): S25-S30.
[149] Schindelin J, Arganda-Carreras I, Frise E, et al. Fiji: An open-source platform for biological-image analysis[J]. Nat Methods, 2012, 9(7): 676-682.
[150] Howse J. OpenCV computer vision with Python[M]. Birmingham: Packt Publishing Ltd., 2013.
[151] Zhou C, Liang D, Yang X, et al. Wheat ears counting in field conditions based on multi-feature optimization and TWSVM[J]. Front Plant Sci, 2018, 9: no.1024.
[152] Kong J, Dimitrov M, Yang Y, et al. Accelerating MATLAB image processing toolbox functions on GPUs[C]// Workshop on General Purpose Processing on Graphics Processing Units. ACM, 2010.
[153] van der Walt S, Schönberger J L, Nunez-Iglesias J, et al. Scikit-image: Image processing in Python[J]. PeerJ, 2014, 2: no.e453.
[154] Kozai T. Resource use efficiency of closed plant production system with artificial light: Concept, estimation and application to plant factory[J]. Proc Japan Acad Ser B Phys Biol Sci, 2013, 89: 447-461.
Indoor phenotyping platforms and associated trait measurement: Progress and prospects
Lingxiang Xu1, Jiawei Chen1, Guohui Ding1, Wei Lu2, Yanfeng Ding1, Yan Zhu3,
Ji Zhou1,4*
(1.Plant Phenomics Research Center/China-UK Plant Phenomics Research Center/Jiangsu Collaborative Innovation Center for Modern Crop Production/Collaborative Innovation Center for Modern Crop Production co-sponsored by Province and Ministry, Nanjing Agricultural University, Nanjing 210095, China;
2.Electrical Engineering, College of Engineering, Jiangsu Key Laboratory of Modern Facility Agricultural Technology and Equipment Engineering, Nanjing Agricultural University, Nanjing 210095, China;
3.National Engineering and Technology Center for Information Agriculture/Ministry of Agriculture and Rural Affairs (MARA) Key Laboratory for Crop System Analysis and Decision Making/Engineering Research Center for Smart Agriculture (Ministry of Education)/Jiangsu Key Laboratory for Information Agriculture, Nanjing Agricultural University, Nanjing 210095, China;
4.Data Sciences, National Institute of Agricultural Botany, Cambridge Crop Research, Cambridge CB3 0LE, Cambridgeshire, UK)
Abstract: Plant phenomics is under rapid development in recent years, a research field that is progressing towards integration, scalability, multi-perspectivity and high-throughput analysis. Through combining remote sensing, Internet of Things (IoT), robotics, computer vision, and artificial intelligence techniques such as machine learning and deep learning, relevant research methodologies, biological applications and theoretical foundation of this research domain have been advancing speedily in recent years. This article first introduces the current trends of plant phenomics and its related progress in China and worldwide. Then, it focuses on discussing the characteristics of indoor phenotyping and phenotypic traits that are suitable for indoor experiments, including yield, quality, and stress related traits such as drought, cold and heat resistance, salt stress, heavy metals, and pests. By connecting key phenotypic traits with important biological questions in yield production, crop quality and Stress-related tolerance, we associated indoor phenotyping hardware with relevant biological applications and their plant model systems, for which a range of indoor phenotyping devices and platforms are listed and categorized according to their throughput, sensor integration, platform size, and applications. Additionally, this article introduces existing data management solutions and analysis software packages that are representative for phenotypic analysis. For example, ISA-Tab and MIAPPE ontology standards for capturing metadata in plant phenotyping experiments, PHIS and CropSight for managing complicated datasets, and Python or MATLAB programming languages for automated image analysis based on libraries such as OpenCV, Scikit-Image, MATLAB Image Processing Toolbox. Finally, due to the importance of extracting meaningful information from big phenotyping datasets, this article pays extra attention to the future development of plant phenomics in China, with suggestions and recommendations for the integration of multi-scale phenotyping data to increase confidence in research outcomes, the cultivation of cross-disciplinary researchers to lead the next-generation plant research, as well as the collaboration between academia and industry to enable world-leading research activities in the near future.
Key words: plant phenomics; indoor phenotyping platform; yield-related traits; quality-related traits; resistance-related phenotypes; phenotyping data management and phenotypic analysis
收稿日期:2020-03-02 修订日期:2020-03-26
基金项目:中央高校基本科研专项资金(JCQY201902);江苏省基础研究计划面上项目(BK20191311)
作者简介:徐凌翔(1984-),男,博士,研究方向:种子活力表型与分析,Email:midway2005@163.com;陈佳玮(1994-),男,硕士,研究方向:表型分析与深度学习, Email:chenjiawei@njau.edu.cn。徐凌翔、陈佳玮对本文有同等贡献,并列第一作者。
* 通讯作者:周 济(1977-),男,博士,教授,研究方向:表型组学、植物形态学、机器学习、图像分析和稻麦育种,电话:025-84395921,Email:Ji.zhou@njau.edu.cn(中),Ji.zhou@niab.com(英)。
doi: 10.12133/j.smartag.2020.2.1.202003-SA002
